
目标区域测序是针对研究者感兴趣的基因组序列,通过定制目标区域的探针,与基因组进行杂交, 将目标区域DNA富集后进行高通量测序。目标区域测序通过对大量样本进行研究,有助于发现和验证疾病相关位点或候选基因。
技术特点
针对目的基因组区域进行遗传变异位点检测,更经济,更高通量,缩短研究周期;对特定区域深入研究,较少数据量就可以获得更深的覆盖度和更高的数据准确性。目标区域测序尤其适用于疾病的大样本量分析。
在标准分析之外,派森诺生物提供多种个性化分析项目。
测序深度
深度推荐至少500X,深度越高所发现的变异信息多。
相关研究
病 种 | 期 刊 | 影响因子 | 时 间 |
扩张型心肌病1 | Science Translational Medicine | 15.843 | 2016 |
乳腺癌2 | New England Journal of Medicine | 55.873 | 2015 |
AML3 | New England Journal of Medicine | 55.873 | 2016 |
实体瘤4 | Clinical Chemistry | 7.911 | 2015 |
慢性淋巴细胞白血病5 | Blood | 10.452 | 2015 |
1、Roberts A M, Ware J S, Herman D S, et al. Integrated allelic, transcriptional, and phenomic dissection of the cardiac effects of titin truncations in health and disease[J]. Science Translational Medicine, 2015, 7(270):270ra6.
2、Easton D F, Pharoah P D, Antoniou A C, et al. Gene-Panel Sequencing and the Prediction of Breast-Cancer Risk[J]. New England Journal of Medicine, 2015, 372(23):2243-57.
3、Papaemmanuil E, Gerstung M, Bullinger L, et al. Genomic Classification and Prognosis in Acute Myeloid Leukemia[J]. N Engl J Med, 2016, 374(23):2209-2221.
4、Chen K, Mericbernstam F, Zhao H, et al. Clinical Actionability Enhanced through Deep Targeted Sequencing of Solid Tumors.[J]. Clinical Chemistry, 2015, 61(3):544.
5、Guièze R, Robbe P, Clifford R, et al. Presence of multiple recurrent mutations confers poor trial outcome of relapsed/refractory CLL[J]. Blood, 2015, 126(18):2110.
1. 基因组DNA:DNA浓度≥20 ng/μl,总量≥2μg; OD 260/280介于1.8-2.0之间,电泳检测无明显RNA条带,基因组条带清晰、完整。DNA无污染、无降解。
2. 血液样品:采集新鲜血液2-3ml于含有EDTA的抗凝管中(紫色盖子),封口处用封口膜密封,样品保存在-20℃冰箱,冰袋运输。
3. 人类组织样本
3.1.新鲜组织:手术新鲜组织黄豆大小2-3个/穿刺组织长度至少1cm,2-3条,置于冷冻管内并于-80℃及以下保存(可保存一年),并于冷冻状态运输;
3.2.蜡块:2年以内标准程序制备的石蜡块,蜡块中组织体积大于1 cm3,室温储存及运输。
3.3.石蜡组织切片-白片:2年以内标准程序制备的石蜡组织切片,未染色的白片,厚度:5-10µm,表面积大于1cm2,10片及以上(如切片厚度或表面积达不到要求需按比例增加切片数量),室温储存及运输。
4. 样品保存期间切忌反复冻融。
分析内容及结果
A: 标准分析 | ||
类别 | 分析内容 | |
1.1 | 数据质控 | |
1.1.1 | 下机数据统计 | |
1.1.2 | 数据质量查看 | |
1.1.3 | 数据获取 | |
1.2 | 序列比对 | |
1.2.1 | 参考序列比对 | |
1.2.2 | 序列比对结果概述 | |
1.2.3 | 目标区域测序深度及覆盖度概述 | |
1.3 | 变异检测 | |
SNP | ||
1.3.1 | SNP检测 | |
1.3.2 | SNP注释及统计 | |
1.3.3 | SNP突变频谱分析 | |
1.3.4 | SNP碱基偏好分析 | |
1.3.5 | SNP在外显子的分布 | |
INDEL | ||
1.3.6 | InDel检测 | |
1.3.7 | InDel注释及统计 | |
1.3.8 | InDel在外显子的分布 | |
1.3.9 | InDel片段长度统计 | |
CNV | ||
1.3.10 | CNV检测,统计 | |
SV | ||
1.3.11 | SV检测,统计 | |
变异位点筛选 | ||
1.3.12 | 高频突变分析 | |
1.3.13 | 突变有害性分析 | |
B: 个性化分析 | |||
类别 | 分析内容 | 备注 | |
2.1 | 多样本变异位点异同分析 | ||
2.1.1 | 多样本分组组间异同分析 | 至少2组,每组≥3个样本 | |
2.1.2 | 多样本独立共有突变筛选 | 样本数≥3 | |
2.2 | 致病候选基因筛查预测 | ||
2.2.1 | 孟德尔遗传病-致病基因分析 | 家系样本采样要求: | |
2.2.2 | 复杂疾病-致病候选基因预测 | 采样要求:单个大家系要求尽可能选取所有的患病样本,多个小家系要求每个家系患病样本数目至少≥3, | |
2.2.3 | 复杂疾病-已知易感基因筛查 | ||
2.3 | 新生突变筛选及分析 | ||
2.3.1 | De novo mutations筛选(DNMs) | 采样要求:对于单个家系:子代+双亲,样本数目至少≥3(成3取样),也可以加上兄弟姐妹的样本(成4取样),多个家系进行研究时候建议对同种疾病(同种亚型)的家系进行成3或成4取样。 | |
2.3.2 | DNMs 频谱分析 | ||
2.3.3 | 新生突变率计算 | ||
2.4 | 候选基因富集分析 | ||
2.4.1 | 候选基因GO功能富集分析 | ||
2.4.2 | 候选基因DO疾病富集分析 | ||
2.6.3 | 候选基因KEGG通路富集分析 | ||
2.5 | 调控网络分析 | ||
2.5.1 | 蛋白互作网络分析(PPI) | ||
2.6 | 肿瘤基因组分析 | 基本要求:Tumor+Normal成对取样,样本数目至少≥2,样本信息,疾病信息 | |
驱动基因/致病机理分析 | |||
2.6.1 | 肿瘤突变负荷分析(TMB) | 信息要求:样本信息,肿瘤信息 | |
2.6.2 | 肿瘤驱动基因预测 | 样本数目至少≥3,能达到几十或者上百为佳,要求同一癌症类型的成对癌组织或者血液 | |
2.6.3 | 肿瘤已知驱动基因筛查 | ||
易感基因分析 | |||
2.6.4 | 肿瘤易感基因筛查 | 1个或者多个遗传性癌症家系样本,家系内选取多位患者癌组织(或血液)及其它正常组织(或血液) | |
异质性分析 | |||
2.6.5 | 肿瘤杂合性缺失分析 | ||
2.6.6 | 肿瘤纯度分析 | ||
2.6.7 | 肿瘤倍性分析 | ||
2.6.8 | 肿瘤异质性---克隆结构分析 | ||
2.6.9 | 肿瘤异质性---克隆进化分析 | 同一患者不同时间段,不同部位取样,或者多个患者肿瘤组织和正常组织的成对取样 | |
病毒整合分析 | |||
2.6.10 | 病毒序列识别 | ||
2.6.11 | 整合位点/热点检测 | 样本数目至少≥30 ,病毒血清学检测阳性的同一癌种患者,Tumor+Normal成对取样 | |
2.6.12 | 整合机制分析 | ||
2.7 | 个性化图形展示 | ||
2.8 | 临床数据整合分析 | ||
部分分析结果展示
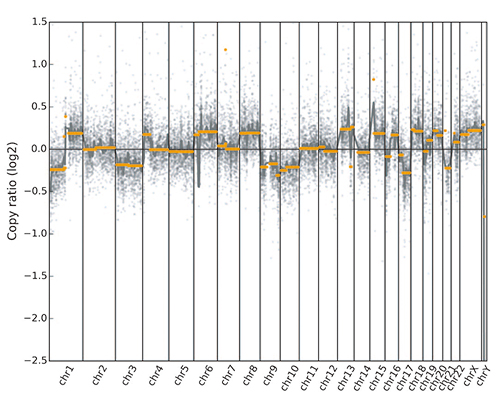
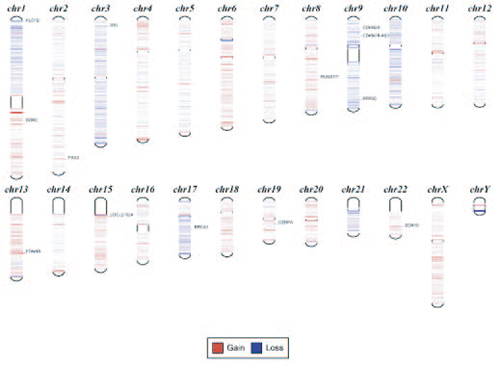
染色体区域拷贝数变异分布
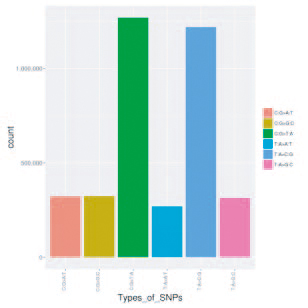
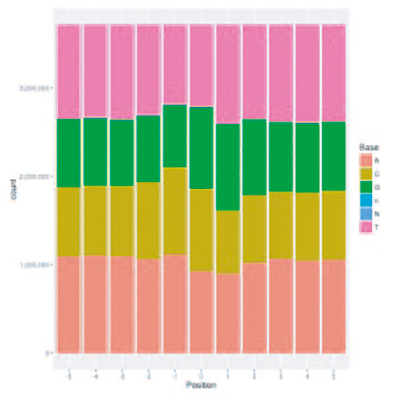
SNP突变频谱分析 突变碱基偏好性分析
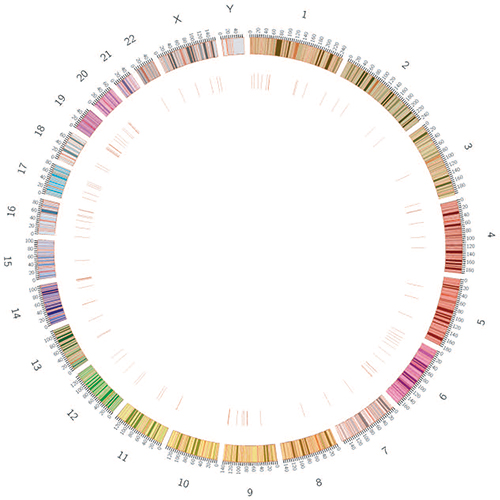
变异位点可视化
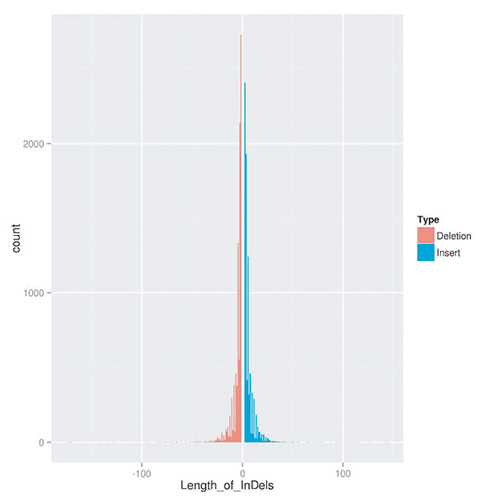
Indel长度分布
Q:与其他测序技术相比,目标区域捕获测序的优势是什么?
A:针对目的基因组区域进行遗传变异位点检测,更经济有效,更高通量,有效缩短研究周期;对特定区域深入研究,较少数据量就可以获得更深的覆盖度和更高的数据准确性。目标区域测序尤其适用于疾病的大样本量分析。
Q:目标区域捕获测序需要客户提供什么信息?
A:客户需要提供需要捕获测序的基因列表或者目标区域所在的染色体号、起点位置、终点位置即可。派森诺会定制相应的探针,并且完成建库、捕获、上机测序到生物信息分析的过程。如果客户自行设计探针,需要在设计之后将ID发送给公司。
- 购买人 会员级别 数量 属性 购买时间
- 商品满意度 :
-








