
真菌近完成图测序
真菌近完成图测序是指对基因组序列未知或无近缘物种基因组信息的某个物种,构建不同插入片段的基因组DNA文库,采用三代单分子测序技术结合二代高通量测序技术对文库进行序列测定,然后利用生物信息学方法进行组装和注释,从而获得该真菌的全基因组和甲基化组。
真菌近完成图测序应用领域
l 获得近完成的真菌全基因组序列图谱
l 获得甲基化修饰相关信息(6mA和4mC)
l 解析真菌基因组中存在的大片段重复序列
技术路线
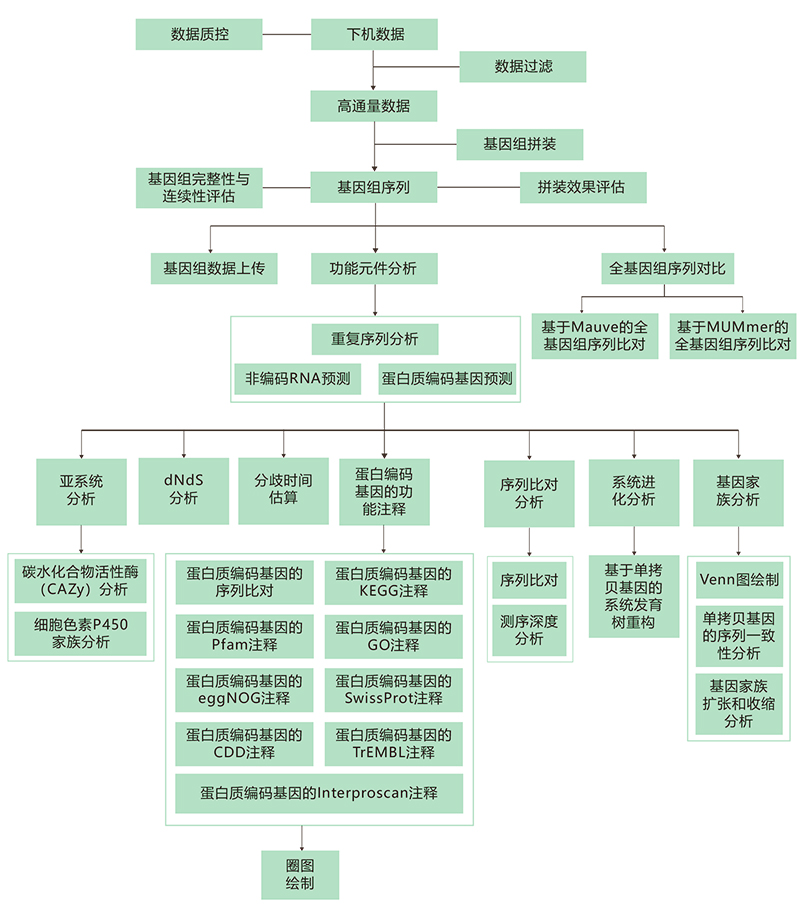
测序送样要求
送样形式 | 送样要求 |
菌体 | 菌体湿重 ≥ 3 g(酵母菌和霉菌) 菌丝球,培养体系 ≥ 1 L,直径1~3 mm(大型真菌) |
DNA | 总量 ≥ 60 µg(荧光定量) OD260/OD280 = 1.8~2.0 OD260/OD230 = 2.0~2.2 |
测序信息分析内容
序号 | 分析项目 | 类型 | 分析 | 备注 |
1 | 下机数据统计 | A | ||
2 | 数据质控 | A | ||
3 | 数据获取 | A | ||
4 | Survey 分析 | A | ||
5 | 基因组拼装 | A | ||
6 | 基因组拼装效果评价 | A | ||
7 | 基因组拼装完整性与连续性评估 | A | ||
8 | 重复序列分析 | A | ||
9 | 非编码 RNA 预测 | A | ||
10 | 蛋白编码基因预测 | A | ||
11 | 碳水化合物活性酶(CAZy)分析 | A | ||
12 | 细胞色素 P450 家族分析 | A | ||
13 | 蛋白编码基因的序列比对 | A | ||
14 | 蛋白编码基因的 GO 注释 | A | ||
15 | 蛋白编码基因的 eggNOG 注释 | A | ||
16 | 蛋白编码基因的 KEGG 注释 | A | ||
17 | 蛋白编码基因的 Swiss-Prot 注释 | A | ||
18 | 蛋白编码基因的结构域分析 | A | ||
19 | 序列比对分析 | A | ||
20 | SNP 分析 | A | ||
21 | InDel 分析 | A | ||
22 | 基因家族分析 | B | 基因组数量 ≥ 2 | |
23 | Venn 绘制 | B | 基因组数量 ≥ 2 | |
24 | 单拷贝基因的蛋白序列一致性分析 | B | 基因组数量 ≥ 3 | |
25 | 基因家族扩张和收缩分析 | B | 基因组数量 ≥ 3 | |
26 | dNdS 分析 | B | 基因组数量 ≥ 2 | |
27 | 基于全基因组单拷贝基因的进化树重构 | B | 基因组数量 ≥ 3 | |
28 | 分歧时间估算 | B | 基因组数量 ≥ 3 | |
29 | 基因组数据上传 | B | ||
30 | 基因组圈图绘制 | A |
A:标准信息分析; B:信息分析。
高级信息分析内容示例
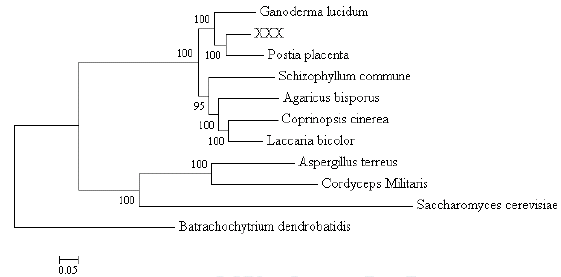
基于单拷贝的全基因组进化分析
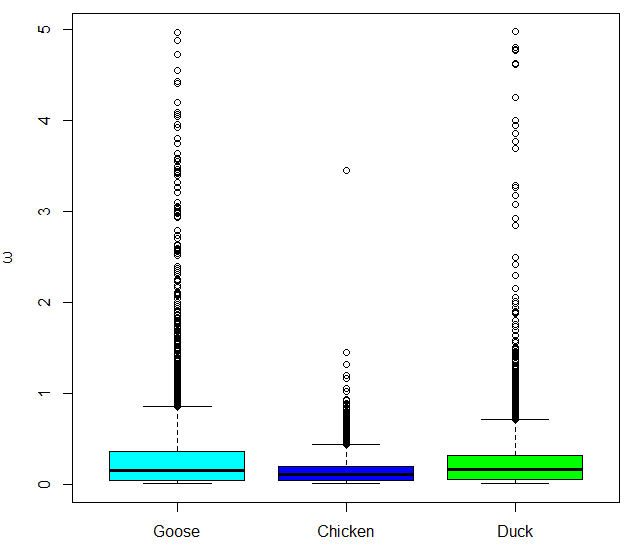
ω值分布图
- 购买人 会员级别 数量 属性 购买时间
- 商品满意度 :
-








