
基因组从头测序(de novo sequencing),主要针对基因组序列未知或参考基因组组装不理想的物种,构建不同类型的基因组DNA文库,并进行序列测定。然后使用生物信息学方法对序列进行拼接、组装和注释,从而绘制该物种完整的基因组序列信息。简单基因组:重复序列低于50%,且二倍体杂合度低于0.5%的物种。
复杂基因组:重复序列高于50%,或二倍体杂合度高于0.5%,或其他多倍体物种。
产品参数
测序方案 | 测序策略 | 交付指标 | 项目周期 | |
简单基因组 | 纯三代 | 文库:20Kb 三代(80X)+10XGenomics/Bionano/ Hi-C | Contig N50≥ 1Mb | (二代)40个自然日(标准分析); |
3+2混合拼接 | 三代(40X)+二代(PE, 100X) | Contig N50≥ 500Kb | ||
复杂基因组 | 纯三代 | 文库:20Kb 三代(100X)+10XGenomics/Bionano/ Hi-C | 视物种而定 | |
3+2混合拼接 | 三代(40X)+二代(PE, 100X) | Contig N50≥ 200Kb | ||
哺乳动物和鸟类 | 纯三代 | 文库:20Kb 三代(80X) | Contig N50≥ 3Mb | |
3+2混合拼接 | 三代(40X)+二代(PE, 100X) | Contig N50≥ 1Mb |
技术路线
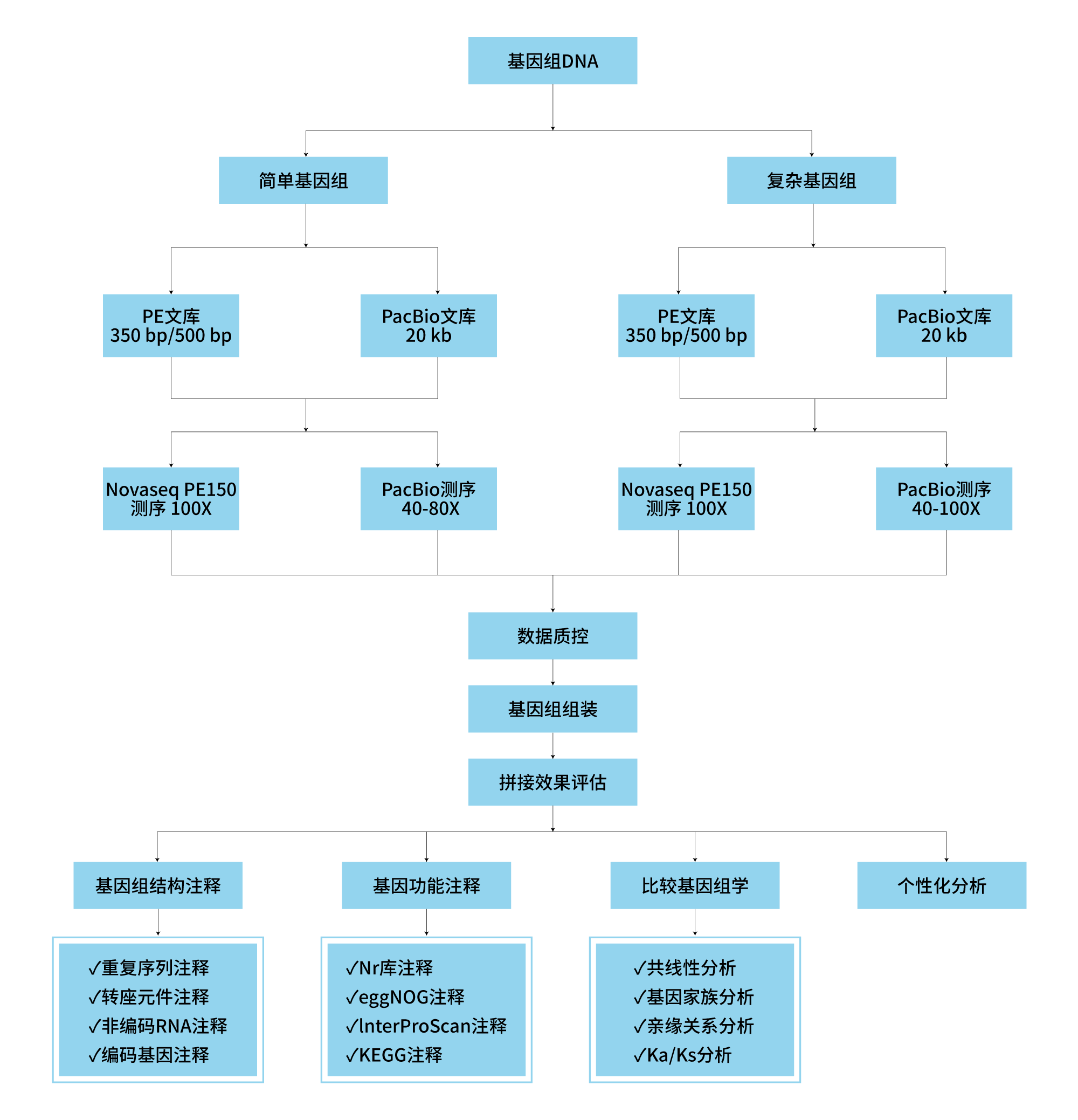
样本要求
| 样 本 | 样本要求 |
|---|---|
| 植物组织 | 嫩叶鲜重2g |
| 高等或大型动物 | 肌肉组织鲜重2g |
| 微体型动物 | 动物体累计鲜重2g |
| 昆 虫 | 正常体型3g,微体型2g |
| 血 液(抗凝) | 正常体型3g,微体型2g |
分析项目
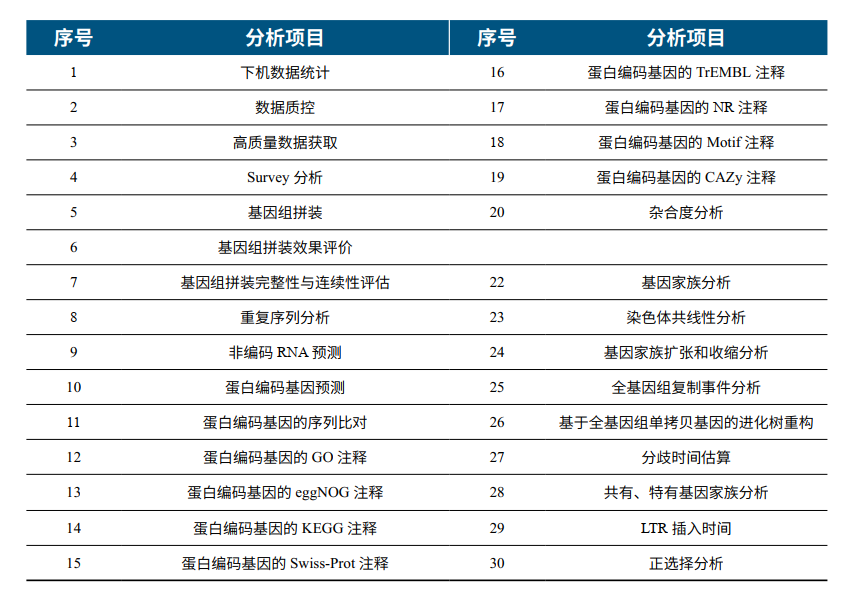
分析结果展示
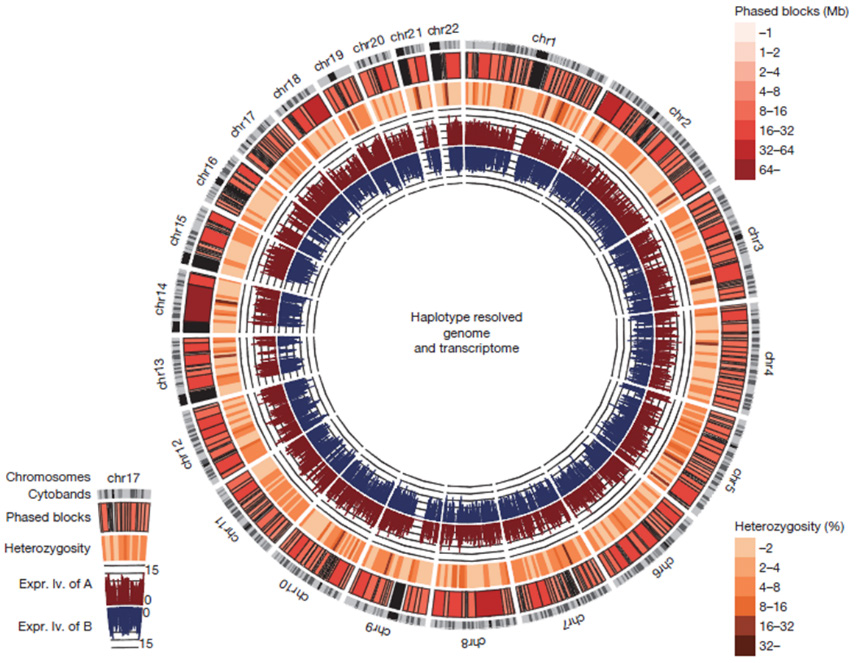
基因组结构与基因表达圈图
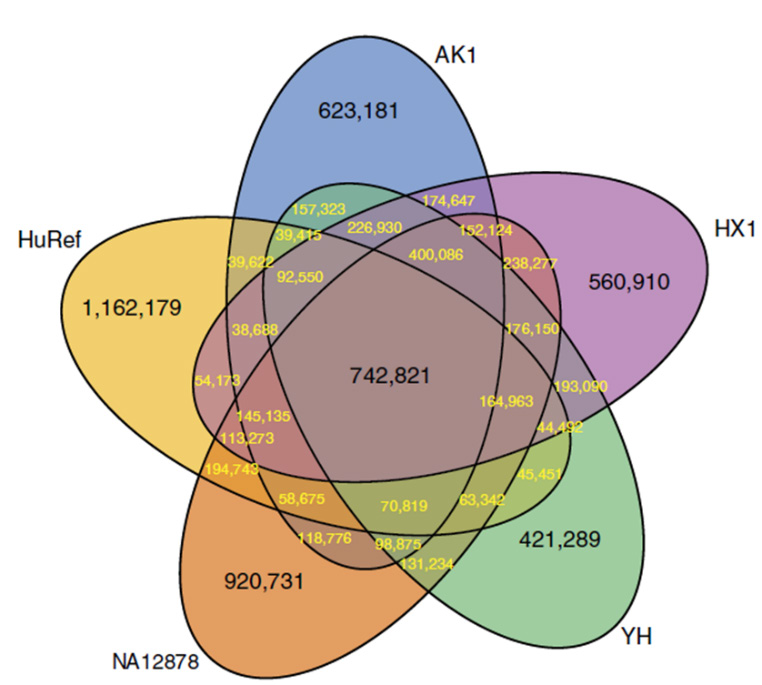
基因家族 Venn 图
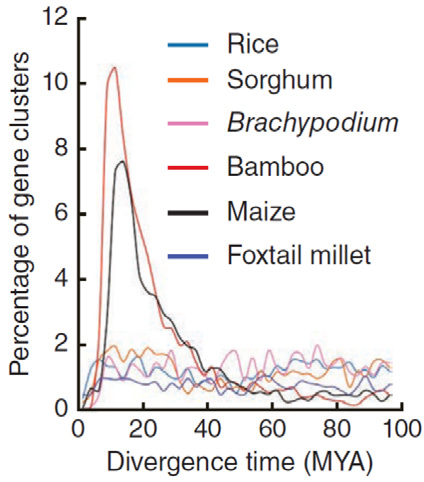
分歧时间估算
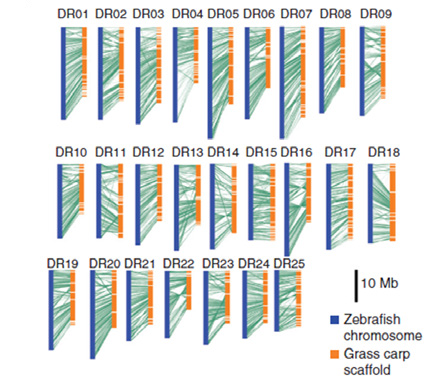
基于基因的共线性分析
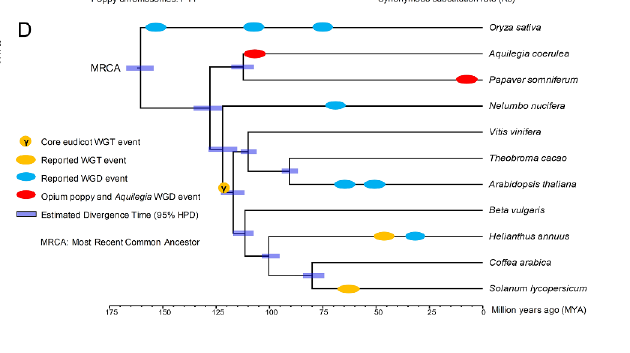
基于全基因单拷贝基因的系统发育树重构
Q:基因组框架图和精细图有什么不同?
A:框架图能覆盖基因组常染色体区域90%,覆盖基因区域95%,Contig N5到5 kb,Scaffold N50达到20 kb,单碱基错误率在十万分之一以下。精细图能覆盖基因组常染色体区域95%,覆盖基因区域98%,Contig N5到20 kb,Scaffold N50达到300 kb,单碱基错误率在十万分之一以下。
Q:动植物基因组从头测序时为什么需要构建不同类型的文库?
A:因为动植物基因组大、复杂度高,且存在大量的重复区域,因此需要制备不同梯度的测序文库,进行双末端测序,使得在拼接中能够跳过大范围的重复区,从而避免了基因组中重复序列造成 的错拼,提高了拼接的质量;同时结合BAC或Fosmid文库以及多种测序平台的综合运用,保证了测序的准确性和基因组的完整性,从而完成高等动植物的基因组图谱绘制。
Q:在动植物基因组从头测序中,不同测序平台分别有什么优势?
A:以Illumina 为代表的二代测序平台,具有测序数据量大,成本低,可以为基因组测序提供高覆盖率的数据的优势。但是该平台存在读长短、具有 GC 偏好性等特点,难以跨过重复序列区域和高 GC 含量的区域。以PacBio 公司的RS II和Sequel 平台为代表的三代测序平台,具有读长长(平均reads 长度 > 10 kb)、测序速度快(运行时间短,单个SMRT Cell的运行时间为2~6 hour)、通量高(平均每个 SMRT Cell 产生~8G有效数据)、无 GC 偏好性、能检测碱基修饰信息(包括甲基化修饰)、无 PCR 偏向性(不需要经过 PCR 扩增,避免测序覆盖的不均一性)等特点,尤其适合动植物基因组的从头测序。根据物种基因组的特点及复杂程度,派森诺生物会科学地设计测序实验方案,合理地将多个平台搭配在一起使用,既能解决复杂基因组的测序难题,保证基因组测序的质量,同时也能兼顾实验项目的经济性。
- 购买人 会员级别 数量 属性 购买时间
- 商品满意度 :
-








