
菌群组成谱全长测序研究
基于PacBio三代测序平台,能够读取群落微生态系统中微生物的rRNA基因/ITS全长序列,不受制于短序列的局限性,并确保全长序列的测序,从而在种甚至菌株等精细水平,解析菌群多样性和组成谱。
产品特色
l 深入获取微生物群落的精细组成信息;
l 预测微生物群落的潜在代谢功能,并指导后续宏基因组测序研究;
l 通过“全微生物组关联研究(Microbiome-Wide Association Studies,MWAS)”,鉴定与表型/组间差异相关的关键物种。
应用领域
l 微生物组与环境互作关系的研究;
l 微生物组与宿主共生关系的研究;
l 微生物组在临床医学和精准医疗领域的应用;
l 微生物组在发酵工艺、食品加工/检测等行业的应用。
赛恒优势
l 自动化、完善的DNA提取扩增体系和样本前处理流程;
l 赛恒拥有PacBio RSII和Sequel两种单分子实时测序平台,提供测序服务;
l Greengenes/Silva/HOMD/RDP/Unite/MaarjAM等多个物种注释数据库,优化物种的分类鉴定;
l 专家教授多方位支持,可按需个性化定制;
l 项目经验丰富,涵盖水样、活性污泥、土壤、排泄物、体液、组织等各类样本。
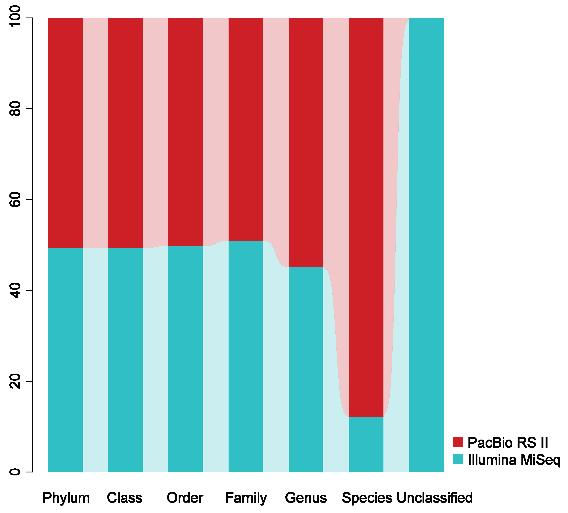

PacBio和Illumina平台对菌群16S rRNA基因测序结果的比较,并改善物种分类信息的不确定性。
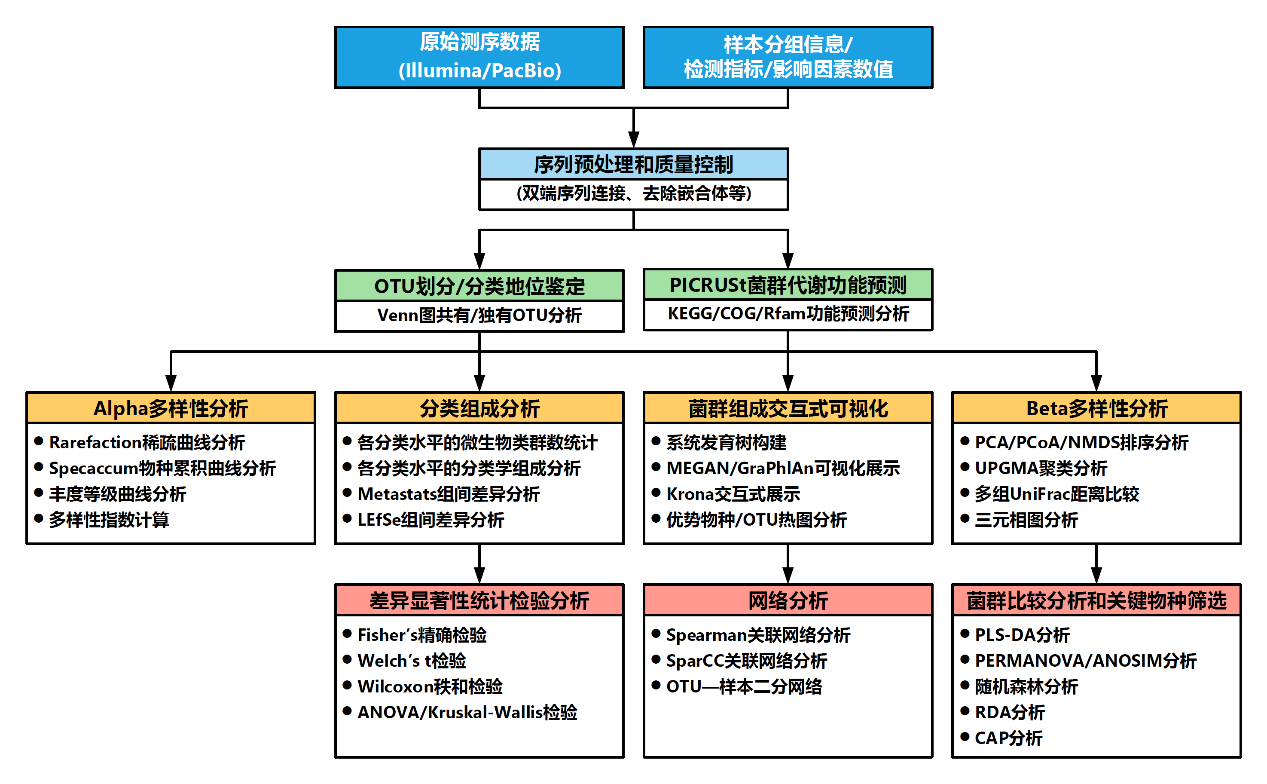
赛恒菌群组成谱全长测序分析流程
送样要求
样本 | 样本要求 |
土壤类 | 500 mg |
排泄物类 | 固体500 mg,液体200 ml |
水样类 | 500 ml液体过滤的滤膜 |
组织 | 200 mg |
微生物基因组总DNA | 体积> 20 ml;浓度> 20 ng/ml |
送样原则:新鲜、足量,请提供三次实验所需用量。
特色分析内容举例
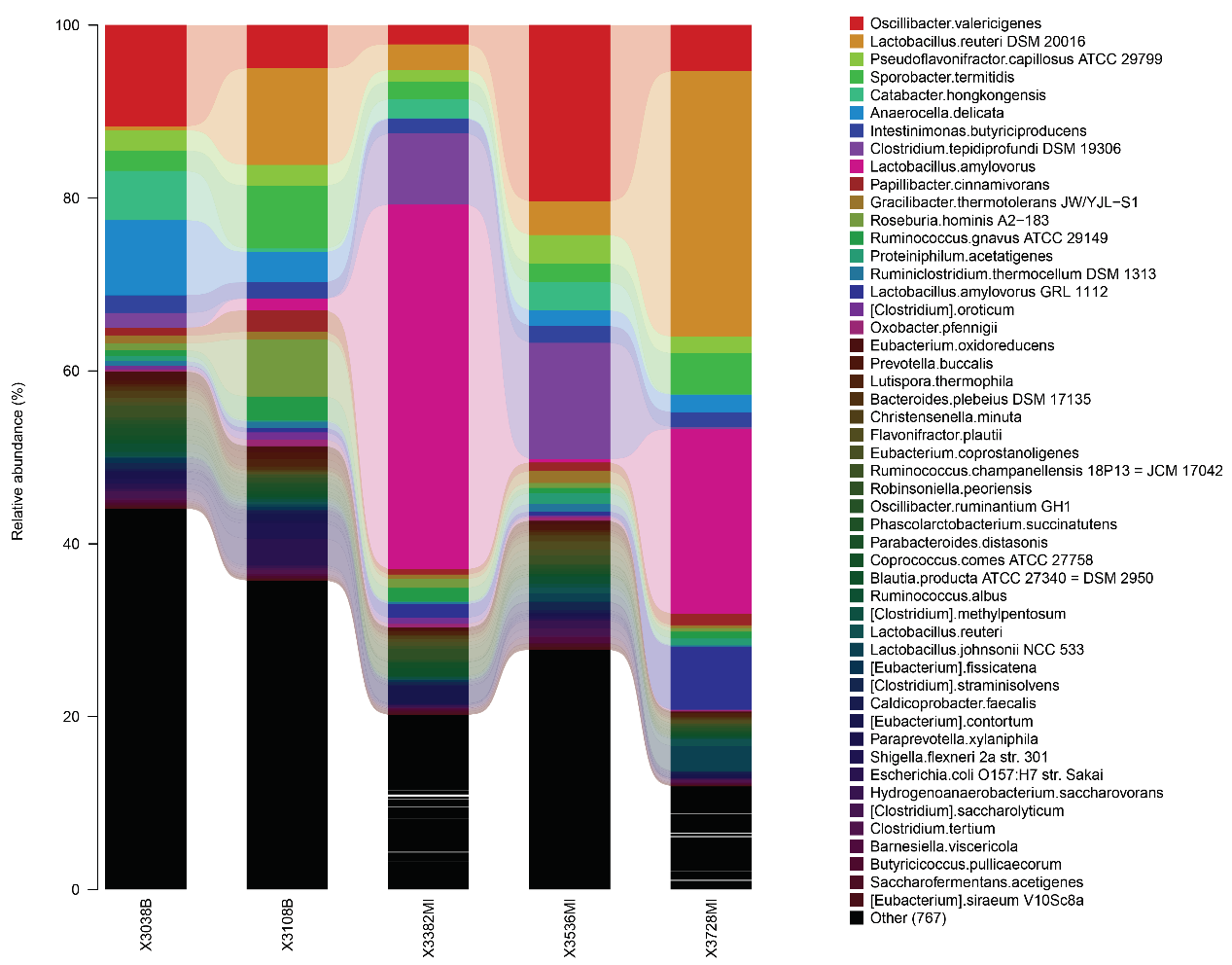
种水平的精细组成图
上图中,横坐标依据样本名排列,每一个柱形图代表一个样本,并以颜色区分各分类单元,纵坐标代表各分类单元的相对丰度,柱子越长,该分类单元在对应样本中的相对丰度越高。
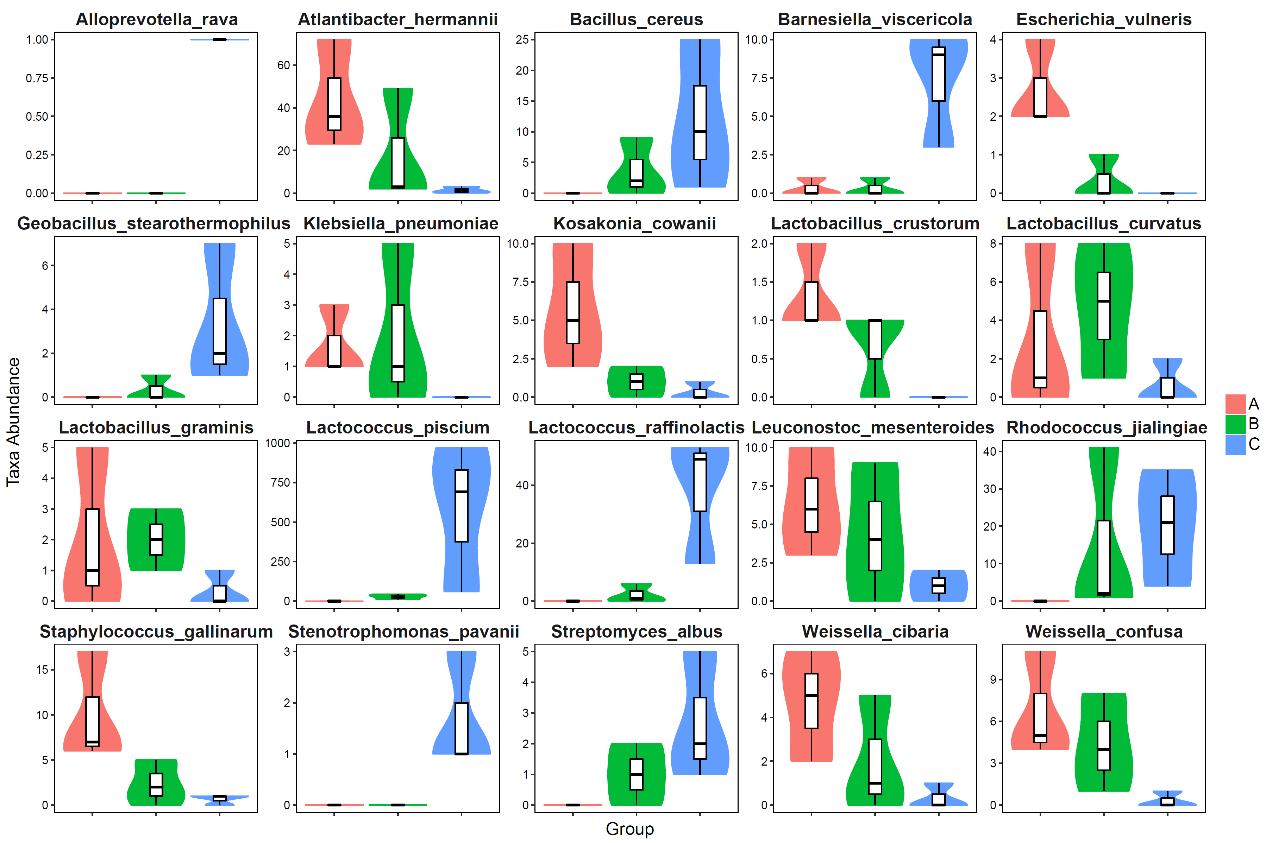
组间差异显著的种水平分类单元的丰度分布小提琴图
上图中,横坐标为组间差异显著的种水平分类单元,纵坐标为各分类单元在各样本(组)内的序列量,以小提琴图结合箱线图的形式展示:其中,小提琴图可以直观地显示数据的分布特征,“小提琴”的“胖瘦”反映了样本数据分布的密度高低(宽度越宽,表明该序列量水平所对应的样本越多);箱线图边框代表上下四分位数间距(Interquartile range,IQR),横线代表中位值,上下触须分别代表上下四分位以外的1.5倍IQR范围,符号“•”表示超过范围的极端值。
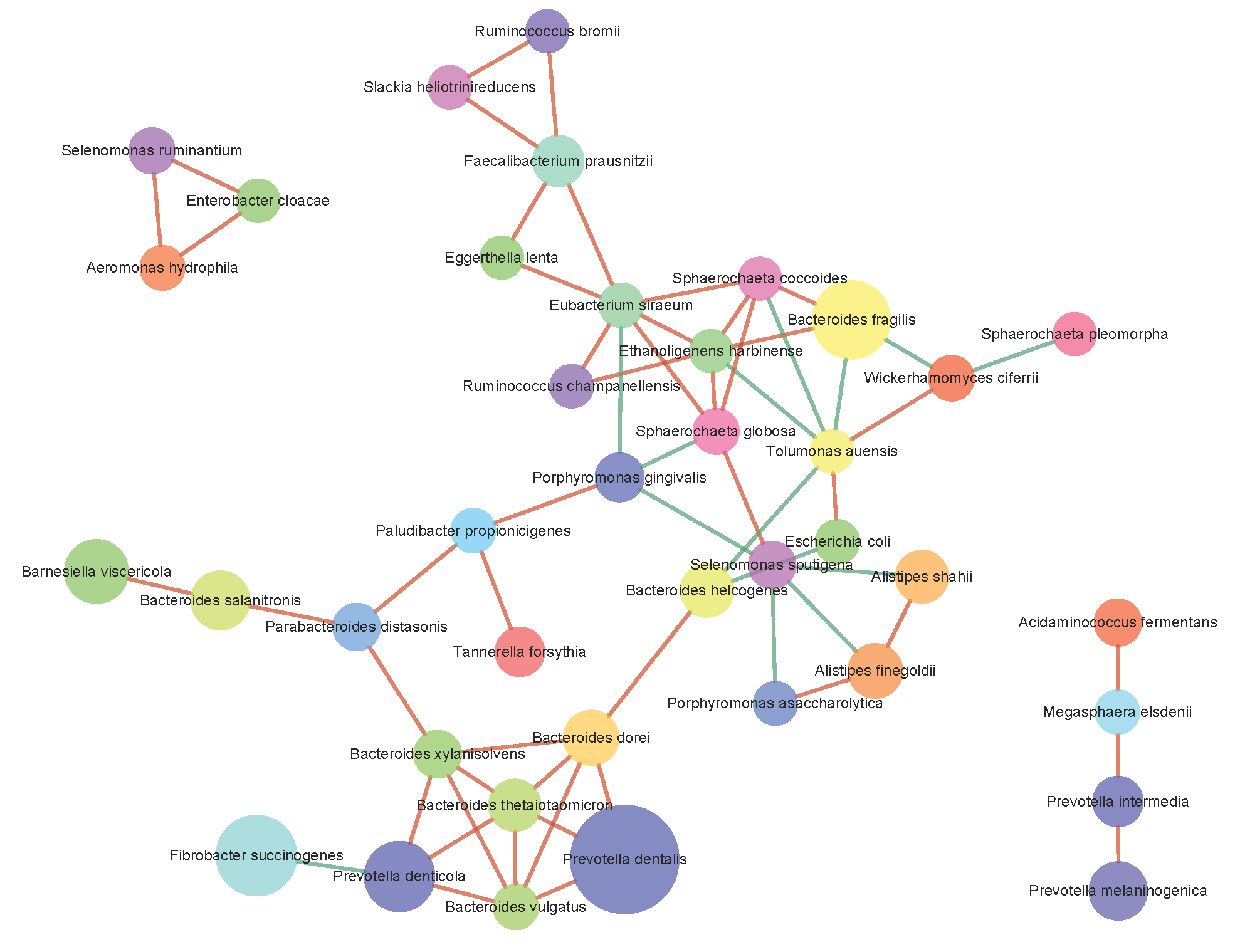
种水平关联网络分析图
上图中,各节点的圆圈代表种水平的各分类单元,以不同的颜色标识,节点之间的连接表明两个物种之间存在相关性,红线表明正相关,绿线表明负相关。通过某节点的连接越多,表明该物种与菌群中其它成员的关联越密切。
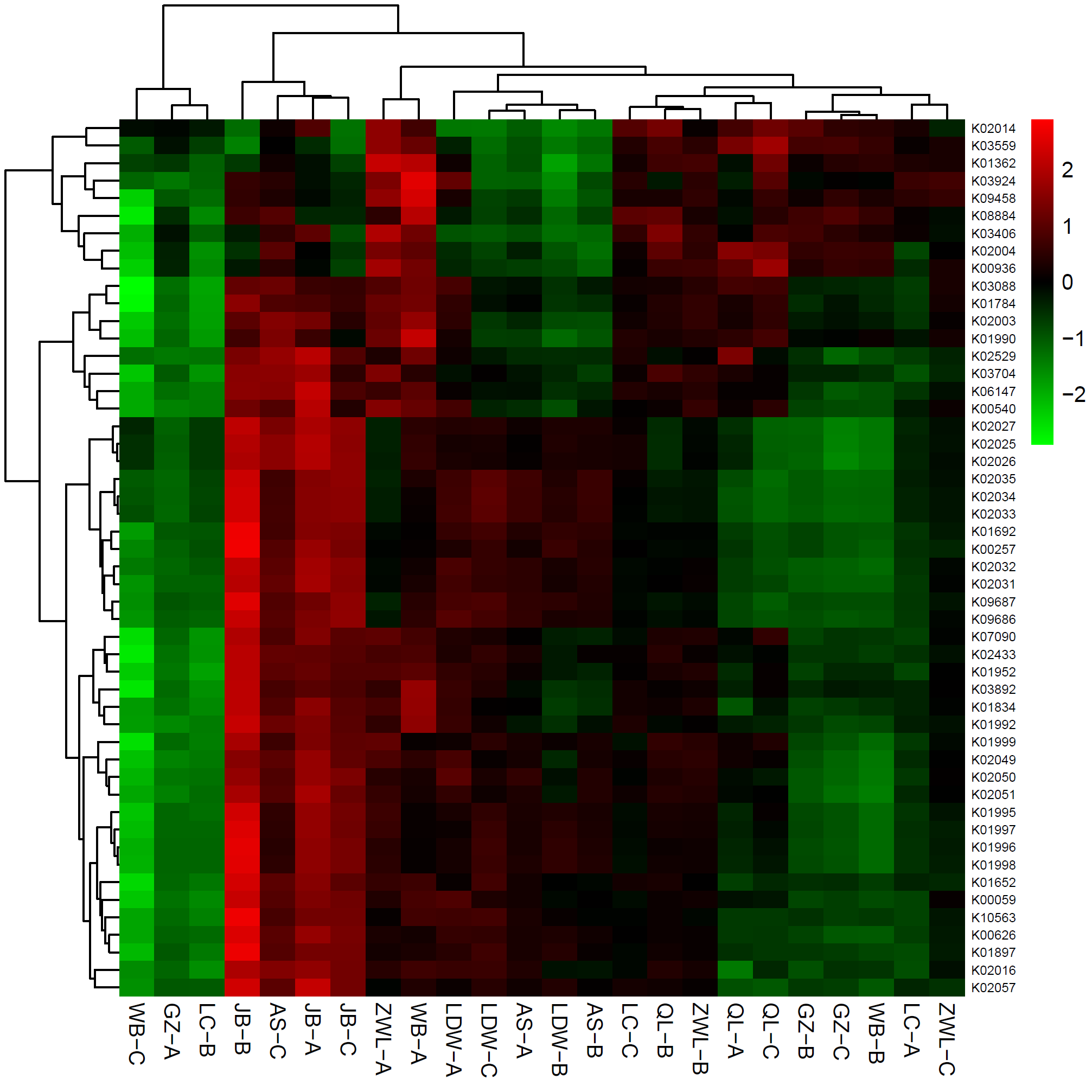
菌群代谢功能预测的KEGG直系同源基因簇(KO)丰度热图
上图中,样本先按照彼此之间功能类群丰度分布的相似度进行聚类,根据聚类结果横向依次排列。同理,功能类群也按照彼此在不同样本中分布的相似度进行聚类,根据聚类结果纵向依次排列。图中,红色代表在对应样本中丰度较高的功能类群,绿色代表丰度较低的功能类群。
- 购买人 会员级别 数量 属性 购买时间
- 商品满意度 :
-








