
细菌全基因组完成图测序
细菌完成图测序是指对基因组序列未知或无近缘物种基因组信息的某个物种,构建不同插入片段的基因组文库,基于 PacBio三代单分子测序技术结合二代高通量测序技术,对这些文库进行序列测定,利用生物信息学方法进行拼接,获得完整的染色体及质粒序列,解析编码信息和表观遗传修饰信息。
应用领域
l 获得完整的染色体序列图谱
l 获得完整的质粒或 BAC 克隆子序列图谱
l 解析甲基化修饰相关信息(6mA和4mC)
赛恒优势
(1) 项目周期短:完成图项目周期 15 工作日,平均项目周期 30 工作日;
(2) 方案设计合理:完成图的方案以三代平台为主、二代平台为辅,得到完整的染色体和质粒序列图谱;
(3) 免费赠送待测菌株的菌种鉴定,以防菌株错测;
(4) 拼接结果准确:三轮校正(拼接前利用三代测序数据间的 Overlap 对测序数据进行校正;拼接后,利用三代测序数据对拼接结果进行第二轮校正;最后,利用高质量的二代测序数据对拼接结果进行多次校正)使得到的基因组拼接结果更加准确;
(5) 项目经验丰富:细菌完成图已完成样品数量近 300 例;
(6) 细节服务到位:无论是圈图起始位点的调整,还是起始密码子的翻译(起始密码子的氨基酸均为甲硫氨酸),力求做到满意。
技术路线
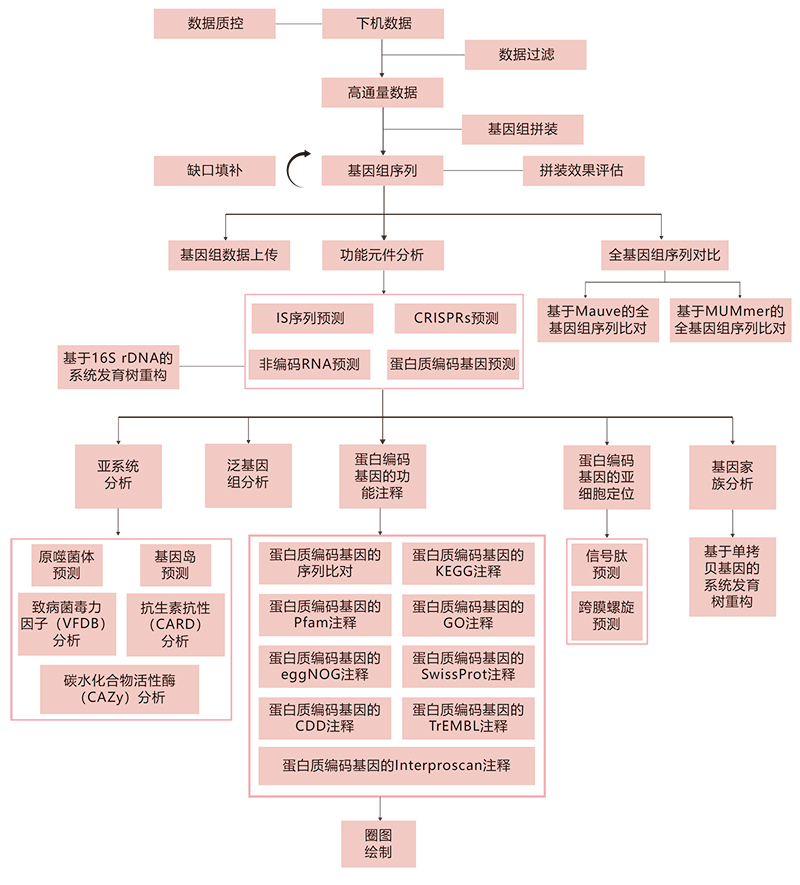
送样要求
送样形式 | 送样要求 |
菌体 | 菌体湿重 ≥ 3 g |
DNA | 总量 ≥ 20 µg(荧光定量); OD260/OD280 = 1.8~2.0; OD260/OD230 = 2.0~2.2 |
信息分析内容
序号 | 分析项目 | 类别 | 分析 | 备注 |
1 | 数据整理 | A | ||
2 | 数据质控 | A | ||
3 | 数据获取 | A | ||
4 | 基因组序列拼装与分析 | A | ||
5 | 蛋白编码基因预测 | A | ||
6 | 非编码 RNA 预测 | A | ||
7 | 其他非编码 RNA 预测 | A | ||
8 | CRISPRs 预测 | A | ||
9 | 原噬菌体预测 | B | ||
10 | 基因岛预测 | B | 完成图 | |
11 | 致病菌毒力因子(VFDB)分析 | B | ||
12 | 抗生素抗性(CARD)分析 | B | ||
13 | 碳水化合物活性酶(CAZy)分析 | B | ||
14 | 信号肽预测 | B | ||
15 | 跨膜螺旋预测 | B | ||
16 | 蛋白编码基因的序列比对 | A | ||
17 | 蛋白编码基因的 GO 注释 | A | ||
18 | 蛋白编码基因的 eggNOG 注释 | A | ||
19 | 蛋白编码基因的 KEGG 注释 | A | ||
20 | 蛋白编码基因的 Swiss-Prot 注释 | A | ||
21 | 蛋白编码基因的 TrEMBL 注释 | C | ||
22 | 蛋白编码基因的 Pfam 注释 | C | ||
23 | 蛋白编码基因的 CDD 注释 | C | ||
24 | 蛋白编码基因的 InterProscan 注释 | C | ||
25 | 基因组圈图绘制 | B | 完成图 | |
26 | 基因组基本信息比较分析 | C | 参考基因组数量≥1 | |
27 | 泛基因组分析 | C | 参考基因组数量≥10 | |
28 | 基因家族分析 | C | 参考基因组数量≥1 | |
29 | 基于 16s rDNA 的系统发育树重构 | C | 参考基因组数量≥2 | |
30 | 基于单拷贝基因的系统发育树重构 | C | 参考基因组数量≥2 | |
31 | 基于 Mauve 的全基因组序列比对 | C | 参考基因组数量≥1 | |
32 | 基于 MUMmer 的全基因组序列比对 | C | 参考基因组数量≥1 | |
33 | 基因组数据上传 | C | 提供相关数据 |
A+B: 完成图标准分析内容; C: 个性化分析内容。
高级信息分析内容示例
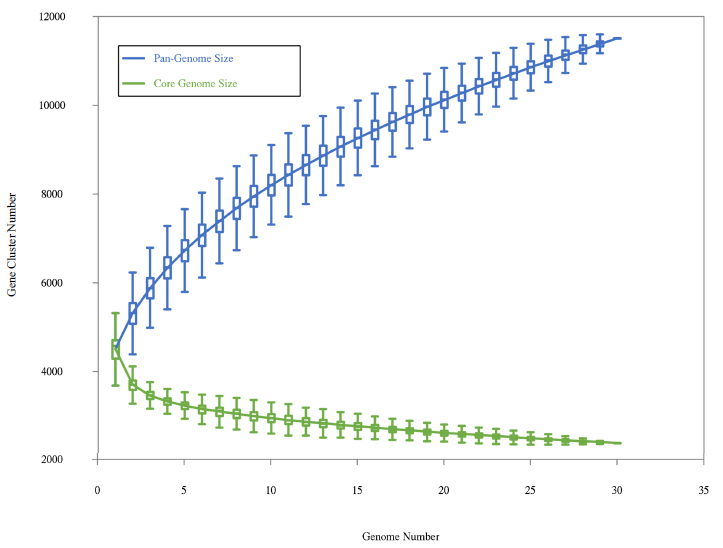
图 1:Pan-Core 基因稀释曲线

图 2 :共线性分析
- 购买人 会员级别 数量 属性 购买时间
- 商品满意度 :
-








