
全长转录组测序
基于PacBio三代测序平台的全长转录组测序,可直接获取mRNA全长序列,更为准确地解读mRNA结构信息,重点关注基因的可变剪切、融合基因等。
测序应用领域
◇ 获得基因组未知物种的基因集,用于基因功能研究
◇ 基因结构差异分析
◇ 发现新转录本
◇ 分子机制研究
◇ 基因表达调控研究
赛恒测序的优势
◇ 完善的样品提取检测方案,保障 mRNA 的完整性;
◇ 赛恒拥有 PacBio Sequel 及 PacBio RSII 单分子测序平台,提供测序服务;
◇ 深度测序推荐,挖掘低丰度基因信息;
◇ 分析团队,项目经验丰富,提供个性化分析内容。
全长转录组实验流程
1、样品制备:总RNA提取
2、cDNA合成:Clontech SMARTer PCR cDNA Synthesis Kit
3、文库构建: SMRTbellTM Template Prep Kit 1.0
4、上机测序:Sequel Sequencing Kit 2.0
技术路线
将提取的总RNA进行反转录,合成cDNA并加接头序列,然后将cDNA进行环化处理,并上机测序。

Pacbio 测序实验流程:
1)高质量总RNA的制备;
2)SMARTer PCR cDNA Synthesis Kit (Clontech) 反转录,将 RNA 反转录成 cDNA;
3)文库构建:SMRTbell™ Template Prep Kit 1.0;
4)上机测序:Sequel™ Sequencing Kit 2.0。

特色分析内容举例
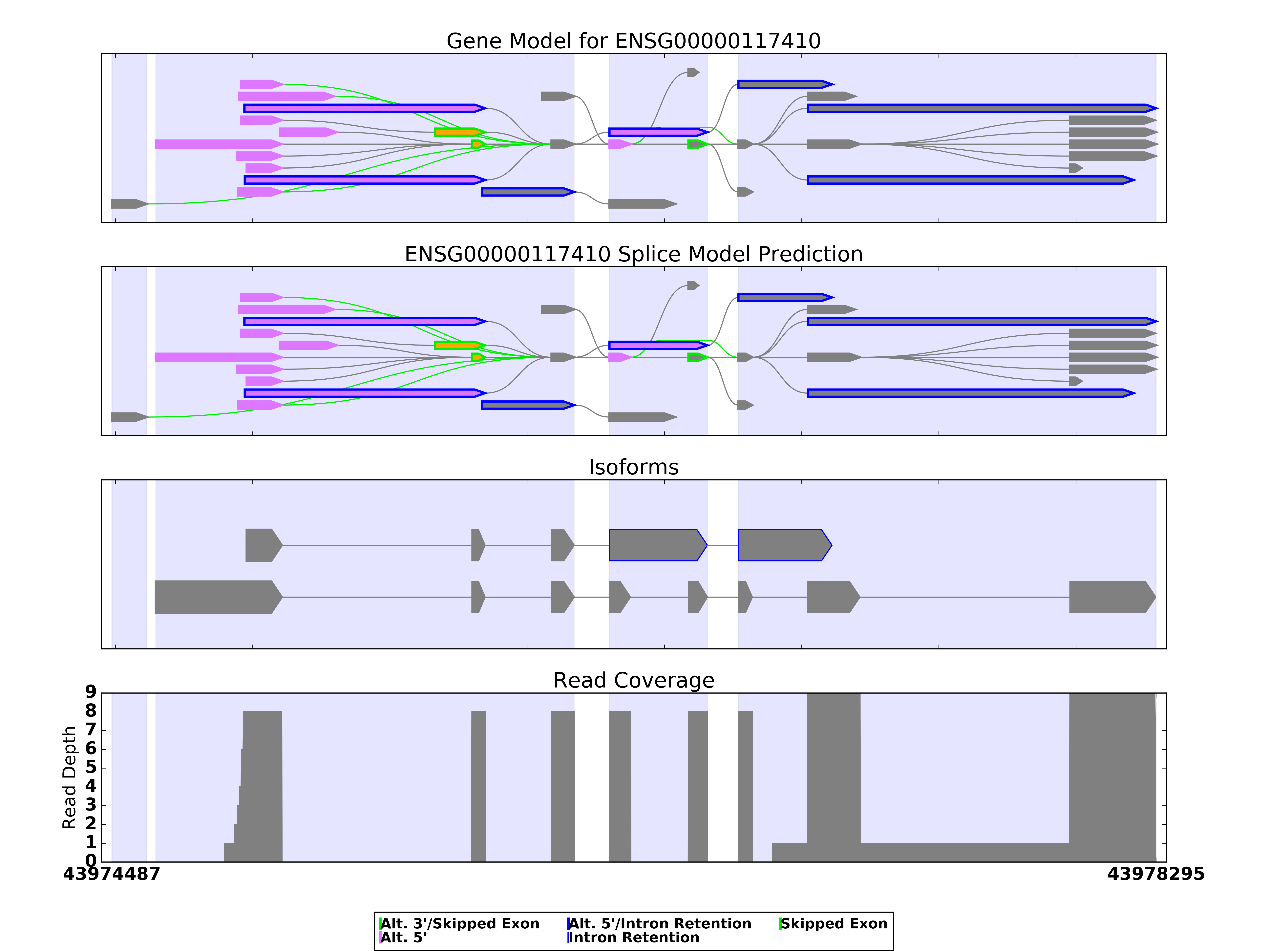
可变剪切模型可视化
上图左下角和右下角的数字分别代表该基因的起始位点和终止位点。该图一共分4行,第一行为以该基因的注释文件作出的基因模型,第二行为根据测序结果与注释文件共同作出的基因模型,第三行也是根据测序结果与注释文件共同作出的基因模型,与第二行不同的是只是画出isoform,第四行则是测序文件中支持各外显子的reads数目。灰色的五边形代表外显子,它们之间的连线表示不同的剪接方式。紫色背景表示有外显子出现的区域(这其中包括保留的内含子),白色背景表示没有外显子出现的区域(即内含子区域)。图中白色背景的宽度并不代表真实的内含子长度,由于有的基因内含子区域远比外显子区域长,为了更清楚地展示可变剪切模型,内含子区域会被缩短很多。
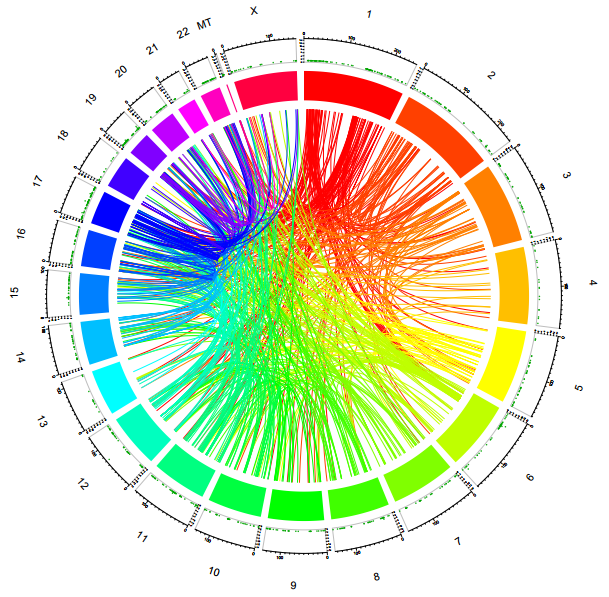
融合基因染色体分布
内圈以连线的形式展示了融合基因的两个组成基因在染色体上的分布位置,外圈则以散点图的形式展示了各位点上的基因成为多少种融合基因的组成成分的情况。
Q:1. PacBio测序平台的技术原理?
A:利用 PacBio RS II/Sequel 进行全长转录组测序,平台包含 16 个测序模块(SMRT cell),每个 SMRT cell 包含 15 万/100万个测序单元(零模波导孔,ZMWs)。需要将 mRNA 进行反转录并进行环化处理,形成 CCS,每个 ZMW(零模波导孔)将容纳 1 个 CCS 分子。由于全长转录组测序对 mRNA 不进行打断,直接进行上机测序,而每个 CCS 的片段长度不同,较小的 CCS 会先落入 ZMWs 中,而导致长片段很难测到。因此,在文库构建时,需要将 mRNA 根据长度分成不同的段,分别构建文库和测序。
Q:2. PacBio测序平台测序方案有什么推荐?
A:对样本进行分段处理,构建不同片段长度的文库,我们推荐每个样本构建3-6个文库。 建议8-15G subreads。
Q:3. 全长转录组测序分析内容包括哪些?
A:分析内容包括:转录本覆盖度计算、转录本功能注释、转录本GO 分类注释、转录本 eggNOG分类注释、转录本KEGG分类注释、ORF分析、可变剪接分析等。
- 购买人 会员级别 数量 属性 购买时间
- 商品满意度 :
-








