
细菌全基因组从头测序(Bacterial Whole Genome de novo Sequencing ),是指对基因组序列未知或无近缘物种基因组信息的某个物种,构建不同插入片段的基因组DNA文库并对文库进行序列测定,然后利用生物信息学方法进行拼接、组装和注释,从而获得该细菌的全基因组序列图谱。
产品参数
产 品 | 文库插入片段大小(bp) | 测序平台及测序模式 | 测序深度 | 交付指标 | 项目周期 |
框架图A | 400 ~ 500 | Illumina MiSeq,PE251 | ~100 × | 数据量 | 30 个自然日 |
框架图B | 400 ~ 500 | Illumina HiSeq,PE150 | ~100 × | 数据量 | 35 个自然日 |
完成图 | 400 ~ 500 | Illumina MiSeq,PE251 | ~100 × | 零 Gap | 45 个自然日 |
20,000 | PacBio | ~100 × |
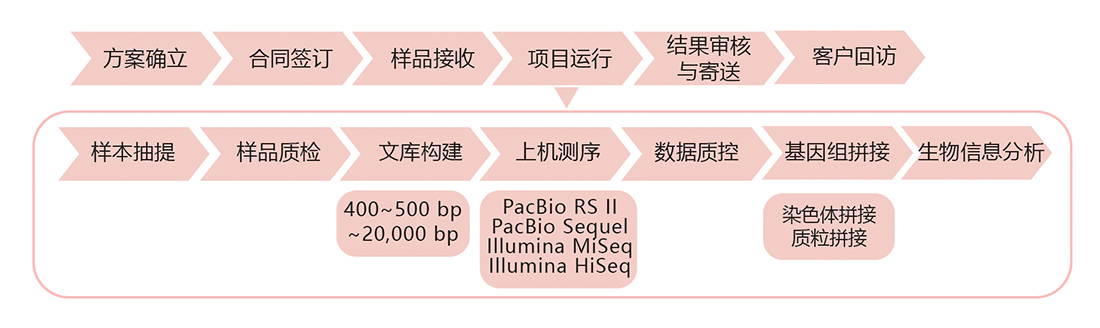
技术路线
信息分析内容
序 号 | 分析项目 | 类 别 | 分 析 | 备 注 |
1 | 数据整理 | A | ||
2 | 数据质控 | A | ||
3 | 高质量数据获取 | A | ||
4 | 基因组序列拼装与分析 | A | ||
5 | 蛋白编码基因预测 | A | ||
6 | 非编码 RNA 预测 | A | ||
7 | 其他非编码 RNA 预测 | A | ||
8 | CRISPRs 预测 | A | ||
9 | 原噬菌体预测 | B | ||
10 | 基因岛预测 | B | ||
11 | 致病菌毒力因子(VFDB)分析 | B | ||
12 | 抗生素抗性(CARD)分析 | B | ||
13 | 碳水化合物活性酶(CAZy)分析 | B | ||
14 | 信号肽预测 | B | ||
15 | 跨膜螺旋预测 | B | ||
16 | 蛋白编码基因的序列比对 | A | ||
17 | 蛋白编码基因的 GO 注释 | A | ||
18 | 蛋白编码基因的 eggNOG 注释 | A | ||
19 | 蛋白编码基因的 KEGG 注释 | A | ||
20 | 蛋白编码基因的 Swiss-Prot 注释 | A | ||
21 | 蛋白编码基因的 TrEMBL 注释 | C | ||
22 | 蛋白编码基因的 Pfam 注释 | C | ||
23 | 蛋白编码基因的 CDD 注释 | C | ||
24 | 蛋白编码基因的 InterProscan 注释 | C | ||
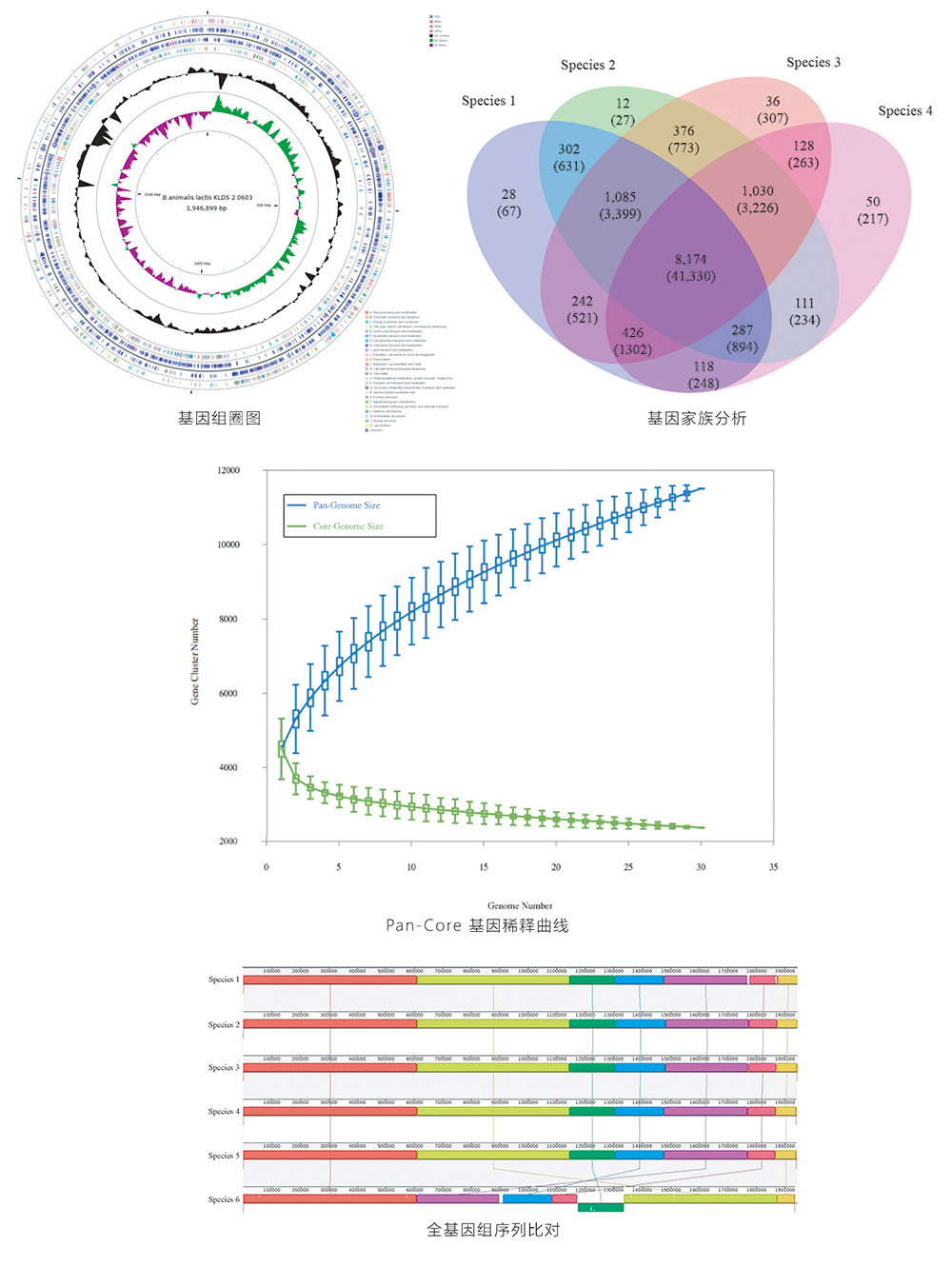
25 | 基因组圈图绘制 | B | ||
26 | 基因组基本信息比较分析 | C | 参考基因组数量 ≥ 1 | |
27 | 泛基因组分析 | C | 参考基因组数量 ≥ 10 | |
28 | 基因家族分析 | C | 参考基因组数量 ≥ 1 | |
29 | 基于 16s rDNA 的系统发育树重构 | C | 参考基因组数量 ≥ 2 | |
30 | 基于单拷贝基因的系统发育树重构 | C | 参考基因组数量 ≥ 2 | |
31 | 基于 Mauve 的全基因组序列比对 | C | 参考基因组数量 ≥ 1 | |
32 | 基于 MUMmer 的全基因组序列比对 | C | 参考基因组数量 ≥ 1 | |
33 | 基因组数据上传 | C | 提供相关数据 |
A:框架图标准信息分析内容; A+B:完成图标准信息分析内容; C:高级信息分析内容。
分析结果展示
- 购买人 会员级别 数量 属性 购买时间
- 商品满意度 :
-








