
全基因组关联分析(Genome-wide association study,GWAS)是一种在某一特定群体中研究遗传突变和表型之间的相关性的方法。通过对遗传多样性丰富的自然群体中的个体进行测序,结合准确的目标性状的表型数据及统计方法进行全基因组分子标记与性状之间的关联分析,可对动植物复杂性状进行定位,快速获得影响目标性状表型变异的遗传标记或候选基因。
产品参数
基于重测序 | 基于简化测序 | |
测序平台 | Illumina Novaseq | |
打断方式 | 物理打断 | 双酶切打断,片段220-450bp |
测序深度 | ≥5×/样本 | ≥2×/样本 |
文库类型 | PE 300~500bp(推荐) | dd-RAD文库 |
样本选择 | 无明显亚群分化的自然群体,个体数不少于200个 | |
DNA样本量 | 总量≥0.5 μg,浓度≥10 ng/μl (单个样本) | 总量≥0.5 μg,浓度≥10 ng/μl (总样本) |
适用范围 | 有参考基因组物种 | 参考基因组较大的物种 |
项目周期 | 标准分析40天 | |
技术路线
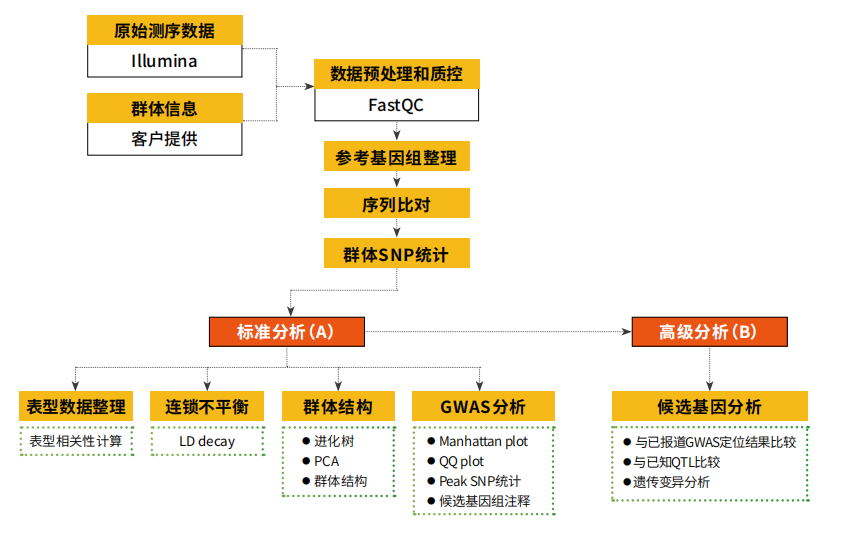
全基因组关联分析主要针对的是自然群体,群体越大,检测效率越高。目前 GWAS 产品建议样本数在 300 个以上。
样本要求 | 测序技术 | 测序量 | 适用范围 |
有参物种的自然群体样本间无亚群分化研究 表型性状遗传力强样本不少于200个 | 重测序(推荐使用) | ≥5X/样本 | 有参考基因组物种 |
简化基因组测序 | ≥2X/样本 | 参考基因组较大的物种 |
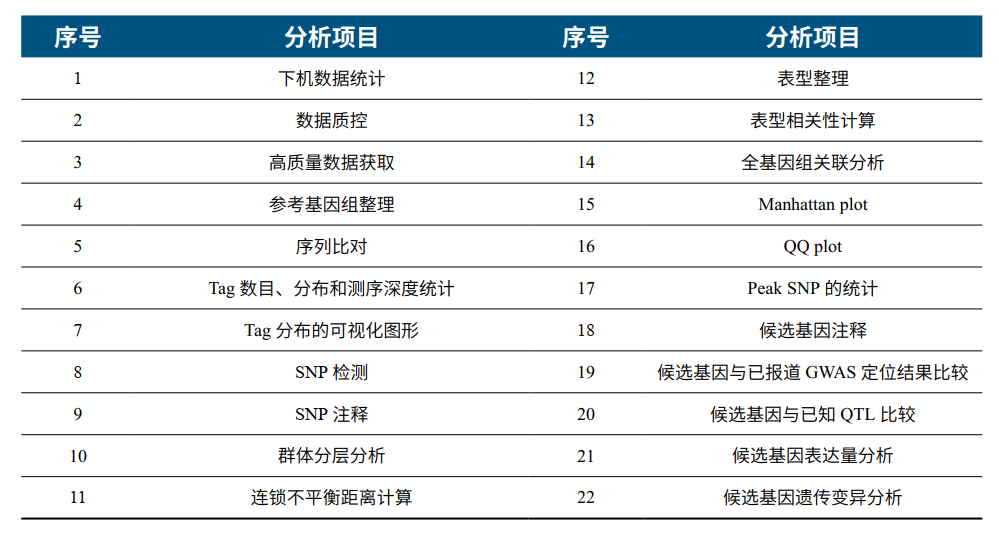
信息分析内容
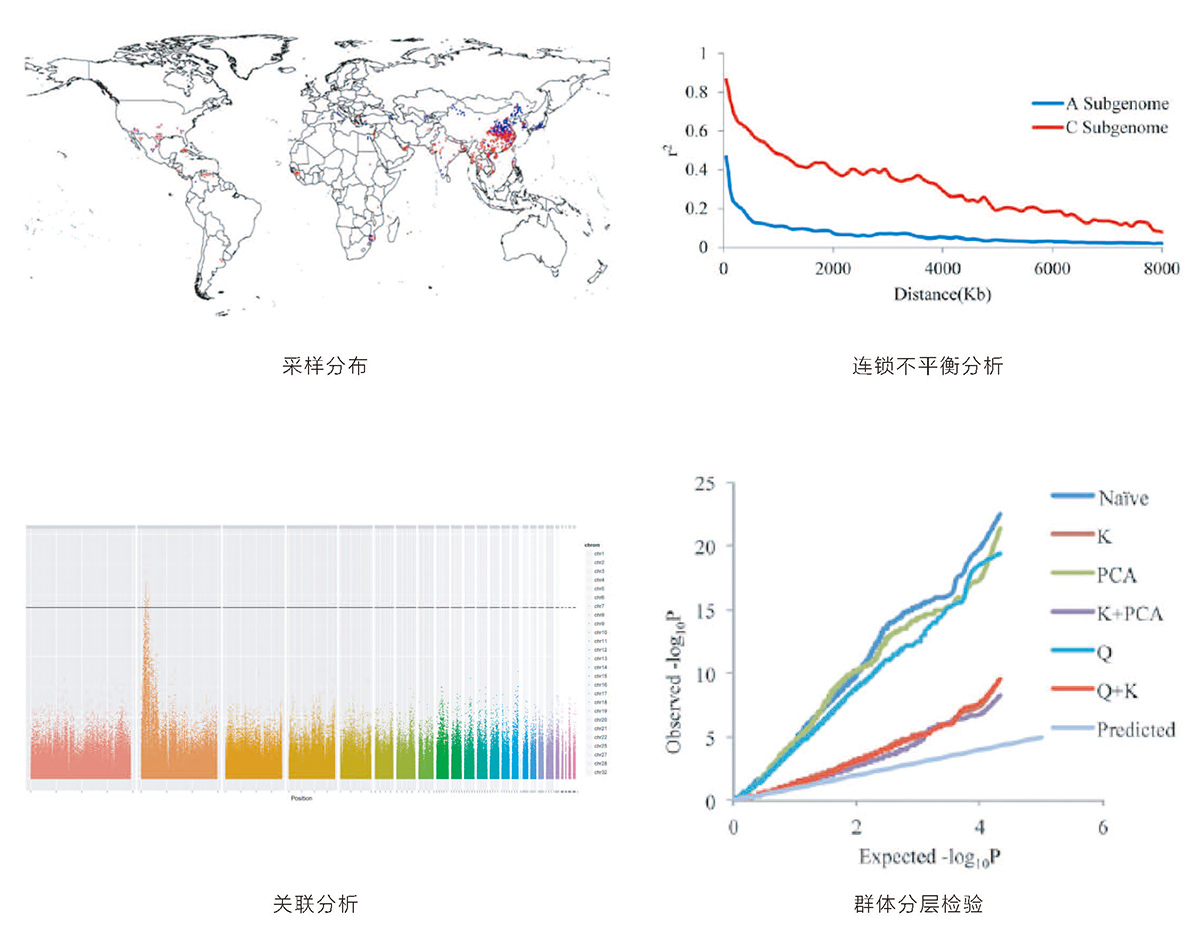
分析结果展示
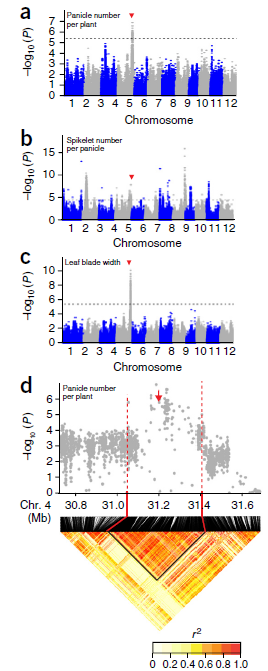
关联区域 LD-block 分析
Wei X, Liu K, Zhang Y, et al. Genetic discovery for oil production and quality in sesame.[J]. Nature
Communications, 2015, 6:8609.
He C, Chen Y, Yang K, et al. Genetic pattern and gene localization of polydactyly in Beijing fatty chicken[J]. Plos One, 2017, 12(5):e0176113.
Xu L, Hu K, Zhang Z, et al. Genome-wide association study reveals the genetic architecture of flowering time in rapeseed (Brassica napus L.).[J]. DNA research : an international journal for rapid publication of reports on genes and genomes, 2016, 23(1):43.
- 购买人 会员级别 数量 属性 购买时间
- 商品满意度 :
-








